近日,实验室团队在《Briefings in Bioinformatics》期刊上发表了题为《DECA: Harnessing Interpretable Transformer Model for Cellular Deconvolution of Chromatin Accessibility Profile》的文章。该研究基于视觉转换器(Vision Transformer,ViT)架构,提出了一种先进的深度学习模型DECA(染色质可及性谱图的解卷积),旨在预测细胞类型的比例,并重建染色质可及性(图1)。
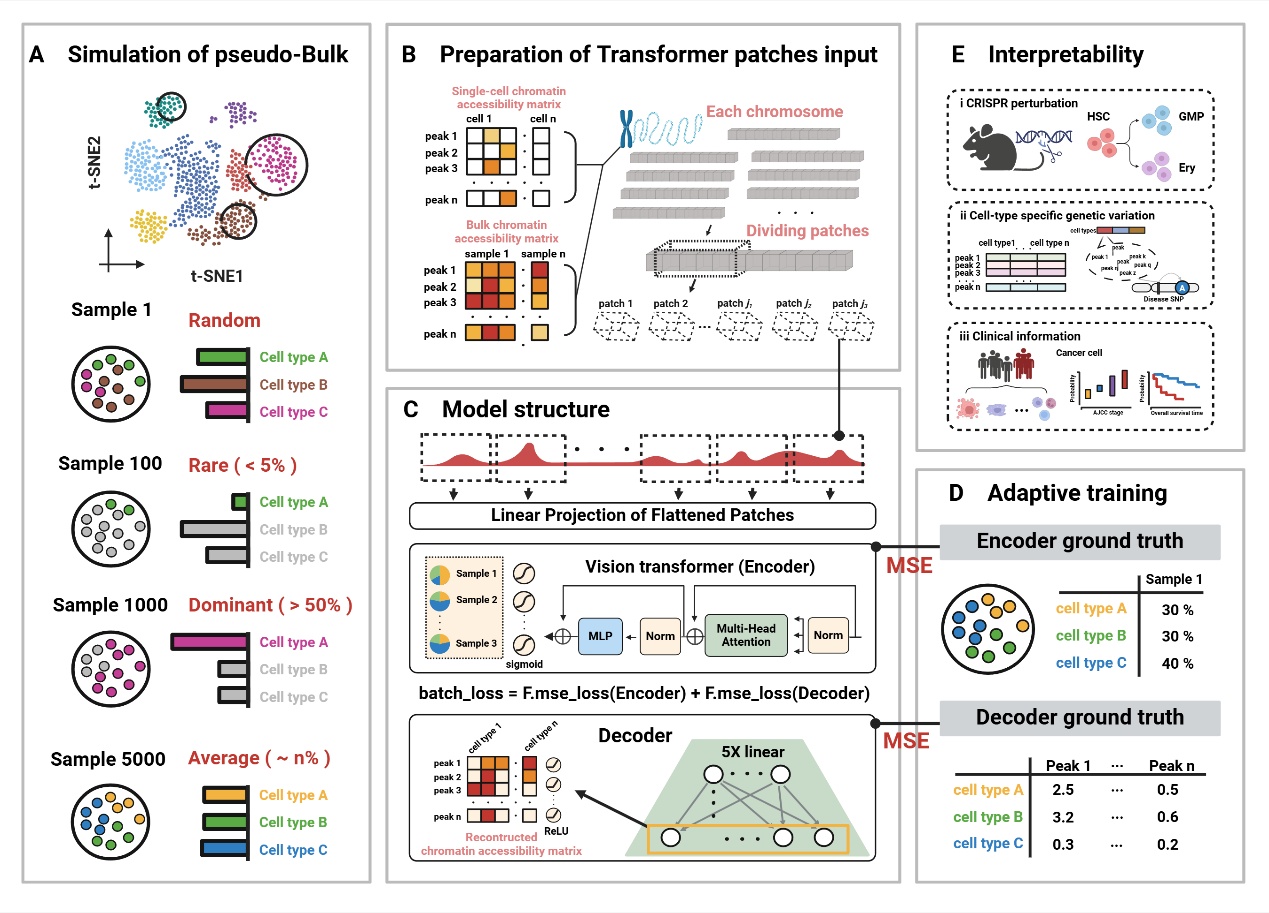
图1. DECA方案流程图
DECA通过将染色体分割为较小的区域(称为patch),并利用多头注意力机制辨识不同patch之间的相互关系。这种方法有效地降低了数据集的维度,同时能够捕捉到与细胞类型特异性高度相关的patch交互(图2)。在DECA的训练阶段,所获得的patch-attention权重帮助从冗余数据中提取关键的染色质共可及性模式。通过基于染色质的层级结构和高分辨率的Hi-C数据,验证了这些经过过滤的注意力权重在生物学上的可解释性(图2)。
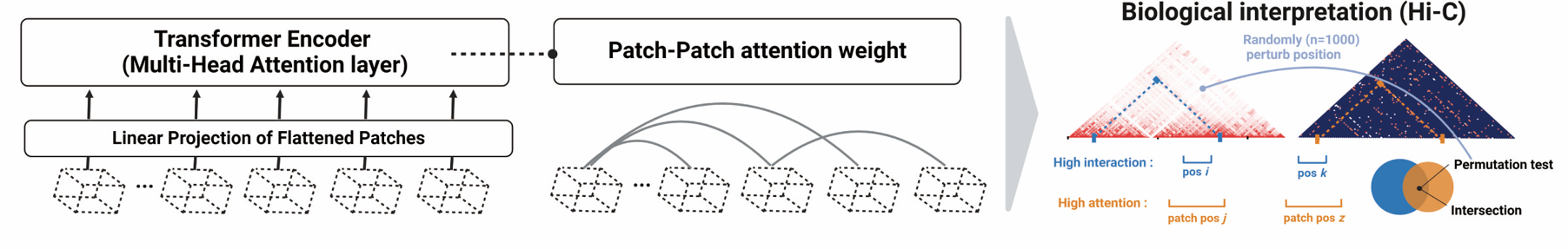
图2. DECA生物学可解释性
该研究由厦门大学健康医疗大数据国家研究院博士生骆世杰,和生命科学学院硕士生林立权完成主要研究工作,厦门大学生命科学学院博士后朱明与黄佳良教授为共同通讯作者。